
RNA 能够执行多种生物功能。
基于计算机技术和人工智能算法,包括 RhoFold、trRosettaRNA、DeepFoldRNA 和 AlphaFold3 等在内的几种基于深度学习的方法已经能够完全自动化地从序列预测 RNA 3D 结构。
近日,麻省理工学院 James Collins 团队开发了一个基于结构预测序列的深度学习平台 RhoDesign,用于 RNA 适配体的从头设计。这种方法可以设计出结构相似但序列不同的 RNA 适配体,能够在小分子存在下产生荧光。
相关文章以题为“Deep generative design of RNA aptamers using structural predictions”发表在 Nature Computational Science 期刊。James Collins 实验室的博士后 Felix Wong、香港中文大学计算机科学及工程系 Dongchen He 是本研究共同一作。Felix Wong 也是 Integrated Biosciences 的联合创始人,这是一家致力于研发与年龄相关疾病药物的新兴生物技术初创公司。
RNA 适配体因其结构的多样性而具有靶分子广、亲和力高、特异性强等特点;同时,相比传统抗体,RNA 适配体分子量小、易改造修饰、制备方便且无免疫原性。因此,RNA 适配体在基础研究、临床诊断、药物研制等方面展现了广阔的应用前景。
RhoDesign 建立在 RhoFold 基础之上,后者在与其他结构预测模型对比时展现出有前景的性能。RhoFold 是由智峪生科团队主导,并联合港中文和复旦大学团队共同完成的全球第一款全自动的端到端 RNA 3D 结构预测深度学习模型。
首先,研究人员整理了 369,499 个由 RhoFold 预测的 3D RNA 结构,以及从蛋白质数据银行(PDB)中获取的 3,435 个 3D RNA 结构,并使用这些结构及其对应的序列来训练深度学习模型;另外,研究人员额外提供了与 3D 结构对应的预测或真实二级结构以提高准确性。
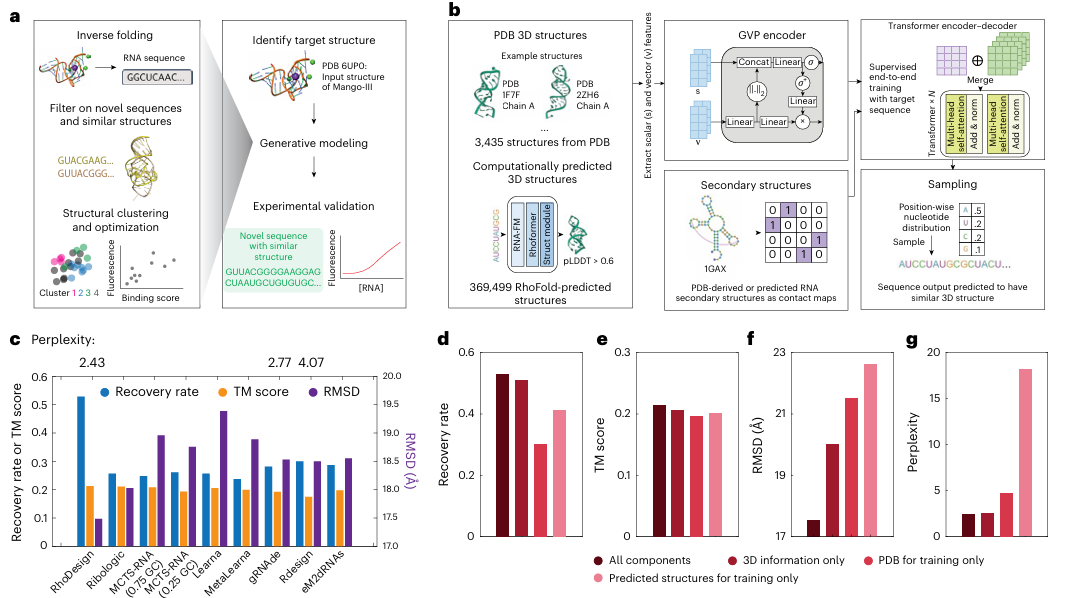
图 | 一种基于 3D 结构的 RNA 生成设计深度学习方法
在模型训练后,研究人员使用恢复率、模板建模(TM)分数、均方根偏差(RMSD)和困惑度来对训练模型进行基准测试。
结果发现,与包括 LEARNA、Meta-LEARNA、RiboLogic、gRNAde、RDesign 和 eM2dRNAs 等在内的模型相比,RhoDesign 的表现更优。
基准测试后,应用 RhoDesign 来从结构生成 RNA 序列。
与小分子 TO1-biotin 结合的 RNA 适配体 Mango 已经被广泛表征。在这里,作者考虑了 PDB 6UP0 中的 Mango-III (A10U)适配体。PDB 6UP0 是 PDB 中的一个条目,它包含了 Mango-III 荧光 RNA 适配体与 YO3-biotin 结合物的三维结构。
首先,他们重新训练 RhoDesign 以排除与 6UP0 中的 Mango-III 适配体序列相似度大于 0.5 的结构,以测试模型的泛化能力;然后,将训练好的模型应用于使用 6UP0 的结构作为输入生成 60 个候选序列,这些序列一致地被 RhoFold 很好地预测。
从生成的序列中,筛选出了 18 个符合条件的适配体进行评估。作为比较,研究人员还分别使用 RaptGen 和其他 7 个从结构到序列生成模型合成了 22 个和 90 个适配体。
在测试合成的适配体在 TO1-biotin(10 微米)存在下的荧光时,发现 18 个 RhoDesign 生成的适配体中有 4 个表现出活性。22 个 RaptGen 生成的适配体中有 20 个也表现出活性,但这些序列与任何 Mango 适配体的最大序列相似度都大于 0.7。
相比之下,4 个活跃的 RhoDesign 生成的适配体与任何 Mango 适配体的最大序列相似度为0.59;其中,适配体 1 更引人注目,它显示出比Mango-I 更高的荧光。从其他 7 个结构到序列生成模型中合成的 70 个适配体中只有 4 个表现出活性;其中,1 个是 gRNAde 生成的,3 个是 MCTS-RNA 生成的。后者的工作命中率为 30%,略高于 RhoDesign 的工作命中率(22%)。

然而,其他方法生成的 4 个活跃适配体都没有显示出比适配体 1 更强的荧光,并且 RhoDesign 在基准测试中显示出大约是 MCTS-RNA 的两倍的恢复率。尽管适配体 1 预测与 Mango-III 结构相似,但它不包含已知的 Mango 适配体荧光活性的基础保守序列基序,这表明它可能是一个前所未有的具有 Mango 样活性的适配体。
为了进一步优化适配体 1,将适配体 1 的 RhoFold 预测结构作为输入提供给 RhoDesign,生成了一组适配体1衍生物。并选择了活性更高的前三名适配体 2-4 进一步研究。
结果发现,适配体 2-4 的荧光强度大于适配体 1。它们的预测 3D 结构,尽管与 Mango-III 的结构相似度比适配体 1 的低,但差异很小,表明它们可能部分通过与 Mango 适配体相似的机制发光。
总而言之,这项研究提供了一个基于计算机的 RNA 序列从头设计平台。与其他平台相比,其可以促进活性 RNA 适配体的结构引导设计,预计将这些方法与 RhoDesign 集成可以进一步提高 RNA 设计的准确性。鉴于当前基准测试的局限性,RhoDesign 也将从使用更可靠的 RNA 3D 结构预测方法进行进一步验证以及未来与多样化 RNA 的额外实验测试中受益。
参考链接:
1.Wong, F., He, D., Krishnan, A. et al. Deep generative design of RNA aptamers using structural predictions. Nat Comput Sci (2024). https://doi.org/10.1038/s43588-024-00720-6






































 浙公网安备33011002015279
浙公网安备33011002015279 本网站未发布麻醉药品、精神药品、医疗用毒性药品、放射性药品、戒毒药品和医疗机构制剂的产品信息
本网站未发布麻醉药品、精神药品、医疗用毒性药品、放射性药品、戒毒药品和医疗机构制剂的产品信息
收藏
登录后参与评论