随着质谱技术的发展,蛋白质组学已成为生物医学研究中的关键工具。然而,如何从庞大的蛋白质组数据中有效提取与疾病相关的生物标志物和关键生物学通路,仍是研究人员面临的重大挑战。传统的分析方法常因其依赖于固定阈值而可能忽略重要的生物学信息,而复杂的深度学习模型尽管提高了分析效率,却常因其“黑箱”性质而缺乏可解释性,限制了在生物医学领域的应用。
来自瑞典隆德大学医学院的Erik Hartman等人提出了一种名为生物信息神经网络(Biologically Informed Neural Networks, BINN)的新方法,用于提高蛋白质组学数据分析的解释性和生物学意义。该研究性论文发表于Nature Communications,题为"Interpreting biologically informed neural networks for enhanced proteomic biomarker discovery and pathway analysis”。
创新方法
BINN的核心思想是利用Reactome数据库所包含有关生物实体关系的信息,生成一个具有生物信息的神经网络模型,其中节点用蛋白质,生物途径或生物过程进行注释,并计算每个特征对模型输出的重要性来解释模型。

Figure 1:利用生物信息神经网络分析蛋白质组学数据的完整工作流程
如图1所示,利用BINN分析蛋白质组学数据的工作流程包括以下步骤:
数据准备:收集患者血浆样本的蛋白质组数据。
BINN构建:利用Reactome通路数据库和蛋白质组数据生成BINN。这一步骤确保了网络结构具有生物学意义。
模型训练:训练BINN区分不同的疾病亚型或严重程度。
网络解释:使用SHAP(SHapley Additive exPlanations)方法解释训练后的网络,识别对分类重要的蛋白质和通路。
生物学洞察: 基于BINN的解释结果,获得疾病机制和潜在生物标志物的新见解。
研究设计与方法
研究团队选择了两个临床相关的疾病数据来验证BINN的效能:脓毒性急性肾损伤(AKI)和COVID-19。(1)对于脓毒性AKI,训练集包含141个样本,其中60个样本被分类为亚型1,81个样本为亚型;测试集包含56个独立样本。(2)对于COVID-19,训练集包含687个样本,其中406个被评为非常严重(WHO量表6-7级),281个评为较轻(WHO量表<6级);测试集包含99个独立样本。在蛋白质组学分析中,AKI数据集鉴定出728个蛋白质,而COVID-19数据集鉴定出173个蛋白质。
研究结果
1. 模型性能比较
为了评估BINN的性能,研究人员将其与其他常用的机器学习方法进行了比较,包括支持向量机(SVM)、K近邻(KNN)、随机森林(RF)、LightGBM和XGBoost。BINN在区分AKI和COVID-19亚型方面表现出色,大大优于传统方法。

Figure 2:机器学习方法在感染性AKI和COVID数据集上的性能
如图2所示,BINN在AKI和COVID-19数据集上均达到了最高的ROC-AUC和PR-AUC值。具体来说:
AKI数据集(图2a-b):ROC-AUC为0.99±0.00,PR-AUC为0.99±0.00
COVID-19数据集(图2d-e):ROC-AUC为0.94±0.01,PR-AUC为0.96±0.01
2. 网络解释与生物学洞察
为了确定对分类较为重要的蛋白质、生物通路和进程,使用SHAP对训练后的BINN模型进行解释。
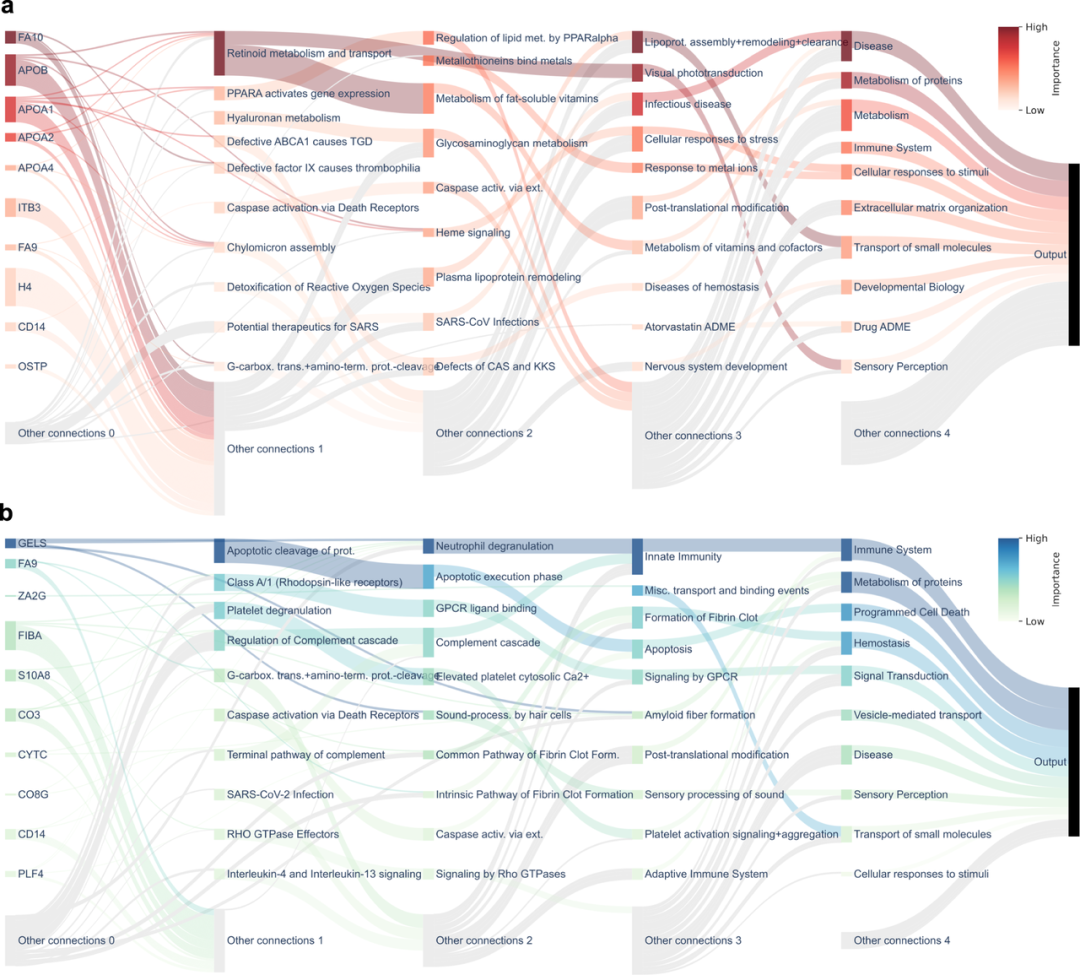
Figure 3:完整脓毒症和COVID-BINN中节点重要性的Sankey图可视化
Sankey(桑基图)是一种用于直观地展示复杂系统中的信息流动的可视化方式。在BINN分析中,桑基图被用来展示网络中不同层级节点的重要性及其连接关系。图3展示了两个Sankey图,分别对应AKI-BINN(图3a)和COVID-BINN(图3b):柱子的大小和颜色深浅反映了其SHAP值。流的宽度表示SHAP值的大小,展示了信息如何从一层传递到下一层。
研究人员发现:
在AKI-BINN中,代谢过程相关的节点(如脂质代谢和PPAR-α相关通路)显示出较高的重要性。
在COVID-BINN中,与免疫系统和细胞死亡相关的节点更为重要。
焕一生物在单细胞分析报告中推出了创新的可解释模块,该模块同样采用桑基图进行可视化。在Reactome图结构的基础上,我们独创性地引入了PubMed文献数量作为权重因子,大大提升了通路分析和结果解读的精确度和可信度,为研究人员提供了更直观、更具生物学意义的结果呈现,助力疾病机制研究和药物靶点发现。
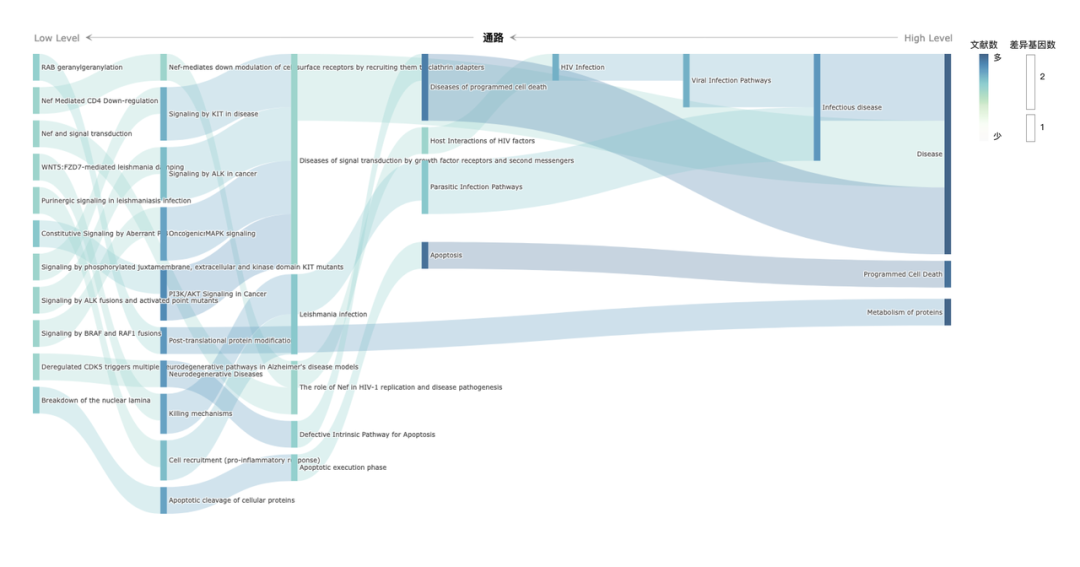
1.图中展示使用差异基因在Reactome中查询到的不同层级的通路,通路层级从左到右依次升高。
2.柱子宽度为关联差异基因数量统计,宽度越高代表通路关联的差异基因越多。
3.柱子颜色代表对应的通路与疾病(Endometriosis)在PubMed中能够检索到的文献数量,颜色越深则代表关联的文献数越多。

差异基因和疾病通路文献关联统计表
3. 增强的生物标志物识别
BINN显著提高了生物标志物的识别能力,不仅能发现已知的标志物,还能识别出潜在的新标志物。

Figure 4:根据SHAP值筛选出脓毒症AKI(上)和COVID(下)中的蛋白质

Supplementary Figure 1:根据差异表达分析筛选出脓毒症AKI(上)和COVID(下)中的蛋白质

Supplementary Figure 2:AKI和COVID数据集差异表达火山图(左)和SHAP值火山图(右)
在急性肾损伤(AKI)数据集中,BINN成功识别出多个关键生物标志物。其中包括已知的炎症标志物如CD14、FA10、H4和OSTP(图4a-b),这些蛋白质在严重脓毒症中表达发生显著变化。特别值得注意的是,可溶性CD14被确认为新生儿早发型和晚发型脓毒症的潜在诊断标志物,展示了BINN在临床诊断标志物发现中的潜力。更重要的是,BINN还发现了一系列与代谢过程相关的蛋白质,如载脂蛋白APOB、APOA1、APOA2和APOA4(图4a-b)。这些蛋白质在传统差异表达分析中可能被忽视,因为它们可能不是差异表达最显著的(附图1 a-b)。在COVID-19数据集中,BINN同样表现出色,成功识别出GELS、ZA2G和S100A8等蛋白质,这些蛋白质已被提议用于区分中度和危重症COVID-19患者,进一步证实了BINN在不同疾病模型中的适用性和有效性。
为了验证BINN方法的优越性,研究团队进行了Rand指数评估。Rand指数是衡量两个数据聚类之间相似性的度量,其值范围从0到1。较高的Rand指数表示两种方法的聚类结果更为一致。结果显示,基于SHAP值识别的蛋白质进行聚类分析,在AKI数据集上达到了0.765的Rand指数,显著优于使用DE评分的聚类(Rand指数0.716)。这种改进意味着BINN方法能够更准确地反映疾病亚型的蛋白质表达模式。在COVID-19分析中,虽然BINN方法和传统差异表达分析的结果较为接近(Rand指数分别为0.663和0.645),但BINN仍然展现出轻微优势。
BINN模型考虑了蛋白质在生物学通路网络中的拓扑位置和蛋白质间的复杂共调控关系,而不仅仅是其表达水平的变化。这种多维度的评估方法使得BINN能够识别出一些在传统差异表达分析中可能被忽视,但在生物学网络中扮演关键角色的蛋白质。这些蛋白质虽然可能不表现出显著的差异表达(附图2),但由于其在通路中的战略位置或与其他重要蛋白质的协同作用,在疾病过程中可能具有重要的调控功能。
4. 深入的通路分析
BINN的结构中包含了各种生物学路径和过程。这种设计使得我们能够从BINN的网络图中提取特定的路径子集,用于进行深入的路径分析。研究人员可以选择一个特定的蛋白质或生物学途径作为起点,然后追踪它与其他途径的相互作用。

Figure 5:利用BINN进行通路分析
图5展示了如何利用BINN进行深入的通路分析: (a) 在AKI-BINN中,CD14主要通过caspase激活和TLR相关疾病通路发挥作用,最终影响疾病、免疫系统和程序性细胞死亡过程(图5a) 。(b) 在AKI中,血浆脂蛋白重塑主要受LDL重塑和几种载脂蛋白(APOB、APOA1、APOA2和APOA4)的影响(图5b)。(c) 在COVID-BINN中,GELS蛋白主要通过影响蛋白质的凋亡裂解和中性粒细胞脱粒,参与程序性细胞死亡和免疫系统调控(图5c)。
5. 跨平台通用性
为了验证BINN的广泛适用性,研究团队还将其应用于使用Olink平台生成的急性呼吸窘迫综合征(ARDS)蛋白质组数据。

Figure 6:由Olink数据训练和构造的BINN
如图6所示,尽管面临小样本量和三分类问题的挑战,BINN仍然能够有效区分健康对照、COVID-19诱导的ARDS和细菌性脓毒症诱导的ARDS(图6b-c),此外,BINN模型还识别出与ARDS相关的几个生物通路,例如G偶联蛋白受体通路(图6a)。以上证明了BINN在不同蛋白质组学平台和复杂临床场景中的适用性。
研究意义
BINN为蛋白质组学数据分析提供了一种新的整合方法,将生物标志物发现、通路分析和机器学习预测模型有机结合。
相比传统的差异表达分析和通路富集分析,BINN考虑了蛋白质丰度、蛋白共表达和通路共调控等信息,有助于发现更多具有生物学意义的结果。
BINN的可解释性使研究人员能够更好地理解复杂生物系统中的分子机制,为精准医疗和药物靶点发现提供新的思路。
生物信息神经网络(BINN)方法的提出为蛋白质组学数据分析开辟了全新的研究方向,也为构建更具生物学意义的可解释AI模型提供了宝贵的启示。焕一生物的单细胞分析报告与BINN方法类似,还引入了创新性可解释模块——采用桑基图结合PubMed文献数量权重。该创新进一步提高了通路分析和结果解读的准确度,助力研究人员在疾病机制研究和药物靶点发现中取得更多突破。未来,焕一生物将继续致力于为科研工作者提供更先进、更精准的分析工具,推动生命科学研究的进一步发展。
关于焕一生物
焕一生物(AliveX Biotech)是中国领先的AI+多组学技术平台公司,拥有端到端干湿实验闭环的多组学实验平台以及AI生物计算平台,包括时空单细胞组学,靶向蛋白组学、代谢/脂质组学等高质量生物检测分析技术以及多组学整合分析、AI建模、知识图谱、疾病药理机制建模等计算技术高效赋能研发。焕一生物拥有国际领先的跨学科院士教授以及产业专家团队,为科研以及药企合作伙伴提供定制化的科研解决方案,包括疾病与药物的机制研究,生物标志物和靶点的发现与评估,患者分层,多模态AI预测,临床试验方案与精准诊疗方案的优化等。焕一生物已服务上百家临床及科研院所和国际大型药企。公司拥有核心技术自主知识产权数十项,并获得国家级高新技术企业认证,美国病理学会CAP认证,ISO9001认证。





































 浙公网安备33011002015279
浙公网安备33011002015279 本网站未发布麻醉药品、精神药品、医疗用毒性药品、放射性药品、戒毒药品和医疗机构制剂的产品信息
本网站未发布麻醉药品、精神药品、医疗用毒性药品、放射性药品、戒毒药品和医疗机构制剂的产品信息
收藏
登录后参与评论